近日,BEVITOR伟德生物数学团队在生物数据建模和统计分析方面取得系列进展。在《Cell》出版社集团旗下的综合性国际期刊《iScience》上发表题为《Exploring Drought-responsive Crucial Genes in Sorghum》(DOI: https://doi.org/10.1016/j.isci.2022.105347)的研究论文。《iScience》在120个综合性(Multidisciplinary)期刊中排名14(14/120)。BEVITOR伟德2020级硕士研究生毕祎琳是论文的第一作者,该工作在王沛教授指导下完成。BEVITOR伟德APP官网是该论文的第一完成单位。

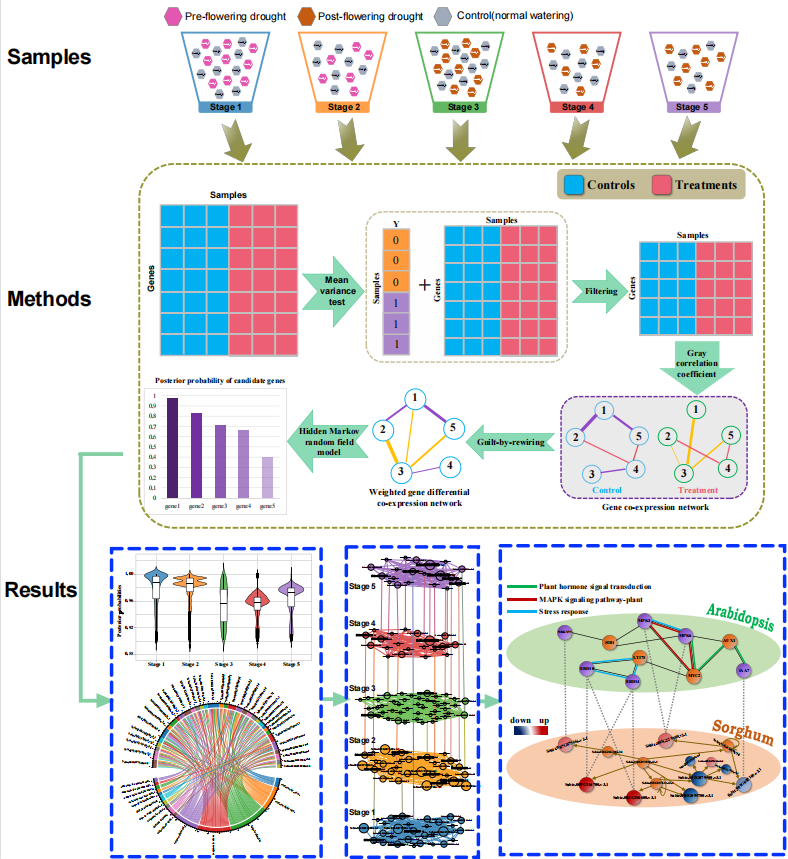
随着全球气候变暖及水资源供需矛盾的加剧,干旱已成为影响全球粮食生产的最主要的非生物因素。高粱是一种典型的耐旱性强的作物。本研究基于多时间点高粱RNA-seq数据,首先通过均方差检验筛选干旱响应的候选基因,构建加权基因差异共表达网络(WGDCNs);然后基于“guilt-by-rewiring”原则,进一步将WGDCNs与隐马尔可夫随机场模型相结合,分别鉴定了高粱的五个发育阶段的干旱响应关键基因。GO富集分析和序列比对分析表明,筛选出的基因可能在高粱的干旱响应胁迫中发挥关键作用。通过构建多层差异共表达网络及其分析,揭示高粱可能对开花前的干旱敏感的网络基础。此外,还建立了一个关键的基因调控模块,该调控模块通过植物激素信号转导、MAPK 级联和转录调控来调节干旱响应。所提出的方法可以很好地通过RNA-seq数据挖掘关键基因,相关研究可以增强我们对高粱干旱响应机制的理解,对基因工程和耐旱新品种的育种具有重要意义。
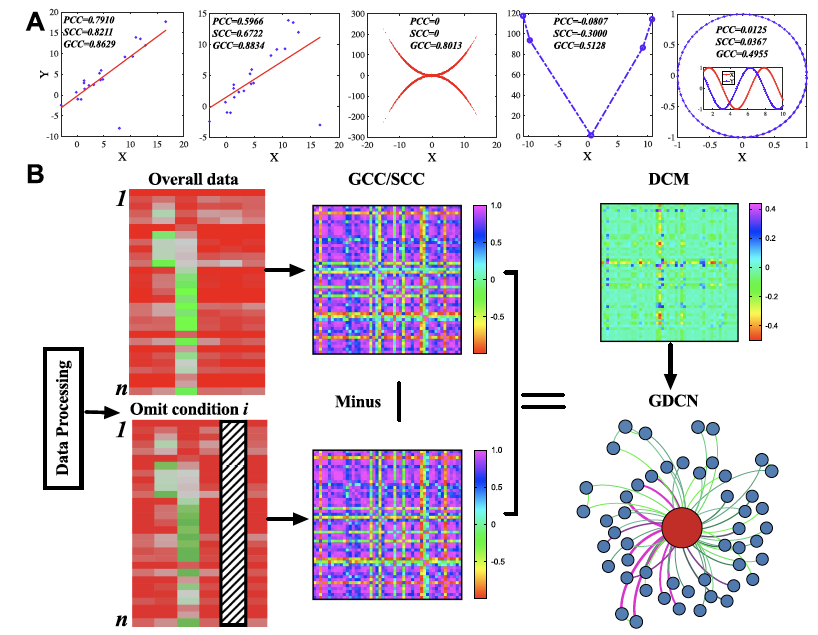
另悉,近期生物数学团队在生物数据建模和分析方面还取得了其他进展。一方面,课题组和农学院王道杰教授合作的论文“Gene differential co-expression networks based on RNA-seq data: Construction and its applications”(https://ieeexplore.ieee.org/document/9512449)于近期在国际期刊IEEE/ACM Transactions on Computational Biology and Bioinformatics上正式发表。该文章提出了针对少样本点转录组数据构建差异共表达网络及利用构建的网络进行生物信息挖掘的框架,并探讨了其在油菜转录组数据中的应用。另一方面,该课题组近期还受邀撰写网络生物学相关进展和惩罚回归模型在生物数据分析方面的进展的综述,对网络生物学的进展以及挑战性问题进行了梳理;对惩罚回归模型在生物数据分析方面的进展和挑战进行了系统总结和展望;进一步明确了课题组未来的研究方向。
在学校的大力支持下,生物数学团队近年来围绕高维生物医学组学数据分析、系统生物学、传染病建模与分析等领域的一些热点问题,在IEEE Trans.系列,Inform Sci.,Nonlinear Dynamics, iScience, BMC Plant Biol.等期刊上取得了系列成果,出版Springer专著1部,主持获得河南省科技进步奖2项等。本研究工作得到了国家自然科学基金、河南省自然科学基金、河南省高校科技创新人才培养计划等项目经费的共同资助。
iScience论文网址:https://www.cell.com/iscience/fulltext/S2589-0042(22)01619-4
IEEE/ACM Transactions on Computational Biology and Bioinformatics论文网址:https://ieeexplore.ieee.org/document/9512449
网络生物学进展及挑战综述论文发表于国际期刊Gene & Protein in Disease,网址:https://accscience.com/journal/GPD/1/2/10.36922/gpd.v1i2.101
惩罚回归模型在生物数据分析中的应用进展的邀请综述论文网址:https://www.mdpi.com/2227-7390/10/19/3695